library(readxl)
library(knitr)
df <- readRDS("df.rds")
df <- df[is.na(df$`BP備考`), ]
dim(df)[1] 584 22ARMS バイオマーカーは質問表応答も予測するか?
司馬博文
6/25/2025
6/25/2025
hirosaki1-3.qmd では BP と \(\Sigma\)-FLC が高いほど項目応答確率が上がることを見た.しかしこれは先行研究 (Wake et al., 2022) と食い違う.2×2 で見たり,交差項を考えたりすることで,この構造をさらに詳しくみる.
元論文のロジスティック解析では,(クレアチン補正した)BP と \(\Sigma\)-FLC の値を正規化せず,そのままのスケールを保つことで discrimination score を構成し,これを cut-off value \(-0.096\) で2値判別することで,AUC \(0.912\) を達成している.
\(\Sigma\)-FLC の備考データも取り除く.
[1] 584 22| id | age | sex | Weight | BP | FLCΣ | BP備考 | 判別式 | LOX | LAB | LOX_Index | type | diversity | BMI | med | med_col | 年代 | 活気 | イライラ感 | 疲労感 | 不安感 | 抑うつ感 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 3 | 4 | 46 | 1 | 76.3 | 1.10 | 1.95 | NA | -2.974 | 136 | 3.8 | 517 | B | 2 | 26.0 | 2 | NA | 40代 | 2 | 3 | 2 | 3 | 2 |
| 5 | 6 | 30 | 1 | 59.5 | 0.76 | 0.64 | NA | -2.776 | 46 | 2.2 | 101 | B | 2 | 19.5 | 2 | NA | 30代 | 3 | 5 | 4 | 5 | 4 |
| 6 | 7 | 51 | 1 | 76.6 | 1.69 | 2.58 | NA | -2.377 | 78 | 3.2 | 250 | B | 2 | 25.5 | 2 | NA | 50代 | 3 | 3 | 3 | 3 | 2 |
| 7 | 8 | 47 | 2 | 43.2 | 0.56 | 0.55 | NA | -3.048 | 40 | 2.7 | 108 | B | 2 | 16.7 | 2 | NA | 40代 | 2 | 1 | 2 | 2 | 1 |
| 9 | 10 | 39 | 1 | 61.9 | 0.52 | 0.64 | NA | -3.165 | 54 | 2.4 | 130 | B | 2 | 21.3 | 2 | NA | 30代 | 3 | 3 | 3 | 3 | 3 |
| 10 | 11 | 55 | 1 | 61.0 | 0.89 | 2.64 | NA | -3.708 | 60 | 3.1 | 186 | E | 1 | 22.9 | 1 | NA | 50代 | 3 | 2 | 2 | 1 | 1 |
に対して長形式のデータは次の通り:
df_IRT_long <- readRDS("df_IRT_long.rds")
df_IRT_long <- df_IRT_long[df_IRT_long$FLCΣ > 0, ]
kable(head(df_IRT_long))| id | BP | FLCΣ | type | diversity | item | response | res | |
|---|---|---|---|---|---|---|---|---|
| 1 | 2 | 0.47 | 0.81 | B | 2 | 活気 | 3 | 0 |
| 2 | 2 | 0.47 | 0.81 | B | 2 | イライラ感 | 3 | 0 |
| 3 | 2 | 0.47 | 0.81 | B | 2 | 疲労感 | 4 | 1 |
| 4 | 2 | 0.47 | 0.81 | B | 2 | 不安感 | 3 | 0 |
| 5 | 2 | 0.47 | 0.81 | B | 2 | 抑うつ感 | 3 | 0 |
| 11 | 4 | 1.10 | 1.95 | B | 2 | 活気 | 2 | 0 |
library(brms)
formula <- bf(
res ~ 1 + (1 | item) + (1 | id) + BP
)
prior_1PL <- prior("normal(0,3)", class="sd", group = "id") + prior("normal(0,3)", class="sd", group = "item")
fit_1PL <- brm(
formula = formula,
data = df_IRT_long,
family = brmsfamily("bernoulli", link = "logit"),
prior = prior_1PL,
chains = 4, cores = 4, iter = 8000
)ran_ef_list <- ranef(fit_1PL)$item[,1,]
ran_id_list <- ranef(fit_1PL)$id[,1,]
df_IRT_long$ran_ef <- ran_ef_list[match(df_IRT_long$item, names(ran_ef_list))]
df_IRT_long$ran_id <- ran_id_list[match(df_IRT_long$id, names(ran_id_list))]
beta_0 <- -2.97
beta_BP <- 0.32
df_IRT_long$discrimination_score <- beta_0 + beta_BP * df_IRT_long$BP + df_IRT_long$ran_ef + df_IRT_long$ran_id
df_IRT_long <- df_IRT_long[!is.na(df_IRT_long$discrimination_score),]
summary(df_IRT_long$discrimination_score) Min. 1st Qu. Median Mean 3rd Qu. Max.
-4.099 -3.726 -3.040 -2.755 -2.035 2.013 param <- seq(-4.1, 2.05, 0.001)
x <- c()
y <- c()
for (threshold in param) {
res_predicted <- df_IRT_long$discrimination_score > threshold
is_false <- res_predicted != df_IRT_long$res
is_true <- res_predicted == df_IRT_long$res
x <- append(x, mean(is_false[df_IRT_long$res == 0], na.rm = TRUE))
y <- append(y, mean(is_true[df_IRT_long$res == 1], na.rm = TRUE))
}
library(ggplot2)
ggplot(data.frame(x = x, y = y), aes(x = x, y = y)) +
geom_point(size = 2) +
geom_line() +
theme_minimal() +
labs(x = "False Positive Rate", y = "True Positive Rate") +
lims(x = c(0, 1), y = c(0, 1))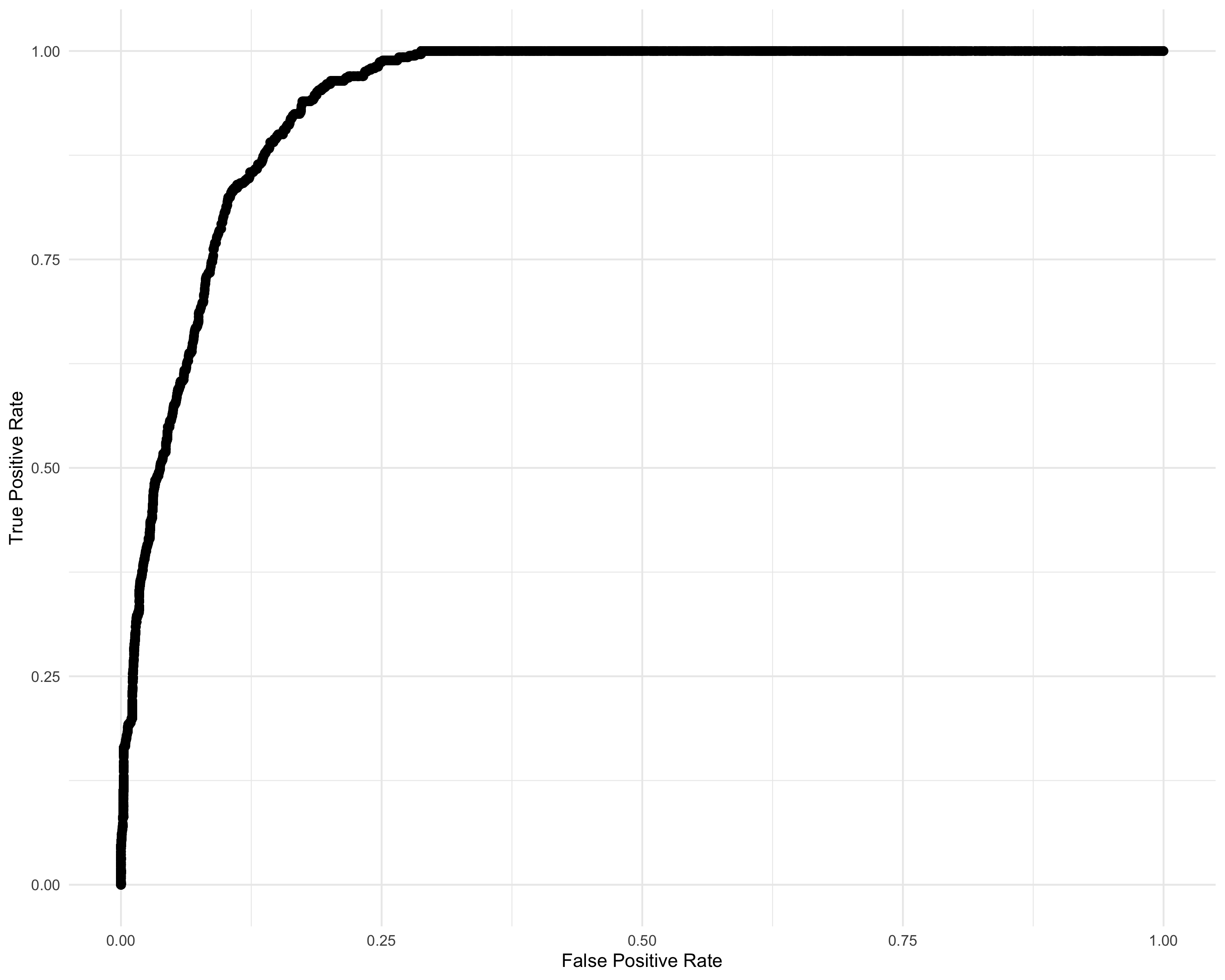
次のパッケージを付け加えます: 'dplyr' 以下のオブジェクトは 'package:stats' からマスクされています:
filter, lag 以下のオブジェクトは 'package:base' からマスクされています:
intersect, setdiff, setequal, unionWarning: パッケージ 'pROC' はバージョン 4.3.3 の R の下で造られましたType 'citation("pROC")' for a citation.
次のパッケージを付け加えます: 'pROC' 以下のオブジェクトは 'package:stats' からマスクされています:
cov, smooth, varSetting levels: control = 0, case = 1Setting direction: controls < cases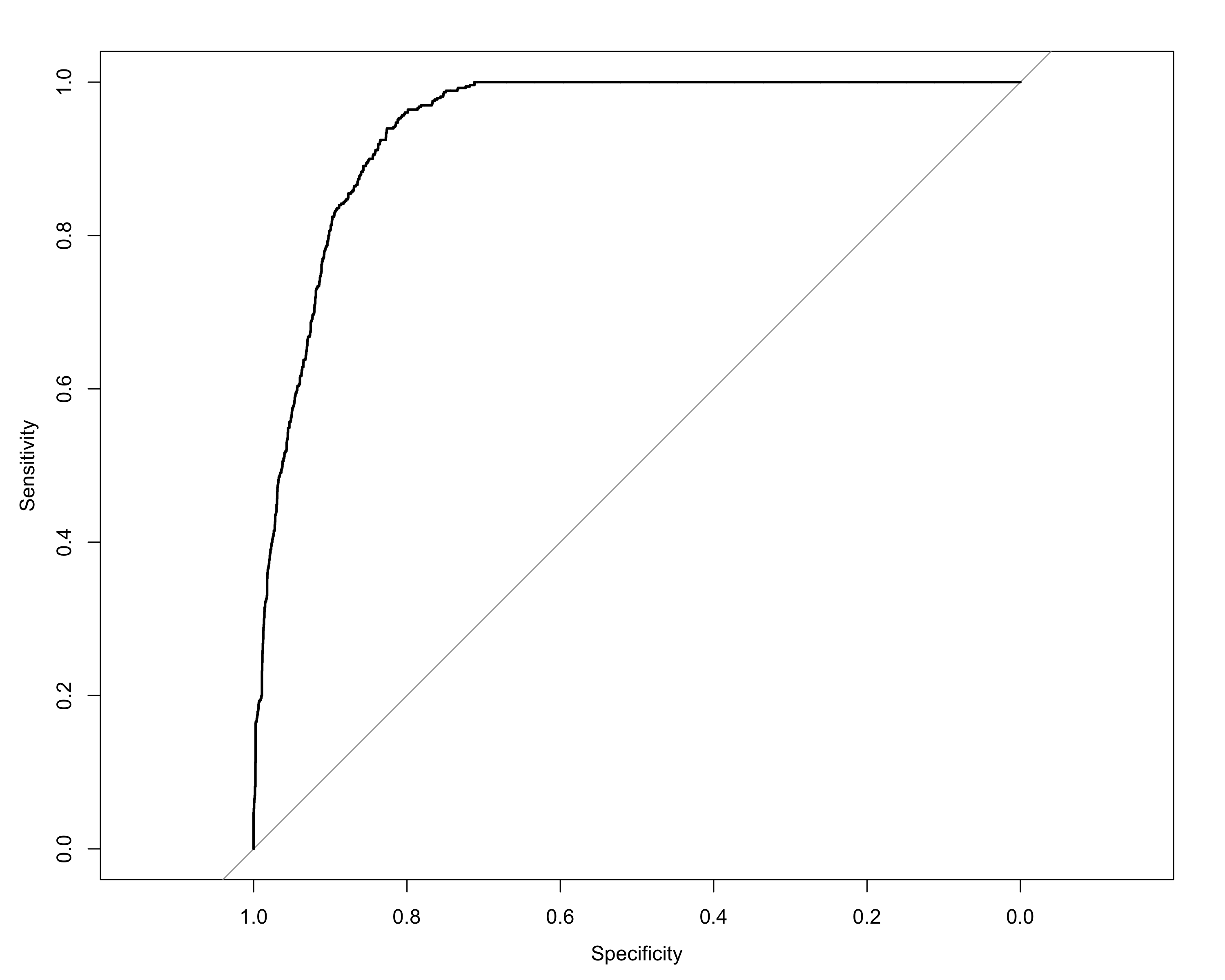
Area under the curve: 0.9409library(ggplot2)
ranef_id <- ranef(fit_1PL)$id
posterior_means <- ranef_id[,1,1]
lower_bounds <- ranef_id[,3,1]
upper_bounds <- ranef_id[,4,1]
plot_df_id <- data.frame(
id = rownames(ranef_id),
mean = posterior_means,
lower = lower_bounds,
upper = upper_bounds
)
plot_df_id <- plot_df_id %>% arrange(mean) %>% mutate(rank = row_number())
p_PL1_id <- ggplot(plot_df_id, aes(x = mean, y = rank)) +
geom_point() +
geom_errorbar(aes(xmin = lower, xmax = upper), width = 0.2) +
theme(
axis.text = element_text(size = 24), # 軸の目盛りの文字サイズ
axis.title = element_text(size = 30), # 軸ラベルの文字サイズ
legend.text = element_text(size = 36), # 凡例のテキストサイズ
legend.title = element_text(size = 24), # 凡例のタイトルサイズ
# plot.margin = margin(r = 20) # 右側の余白を50ポイントに設定
) +
labs(title = "Posterior Means and 95% Credible Intervals for Individuals",
x = "b",
y = "Individual (ID)")
p_PL1_id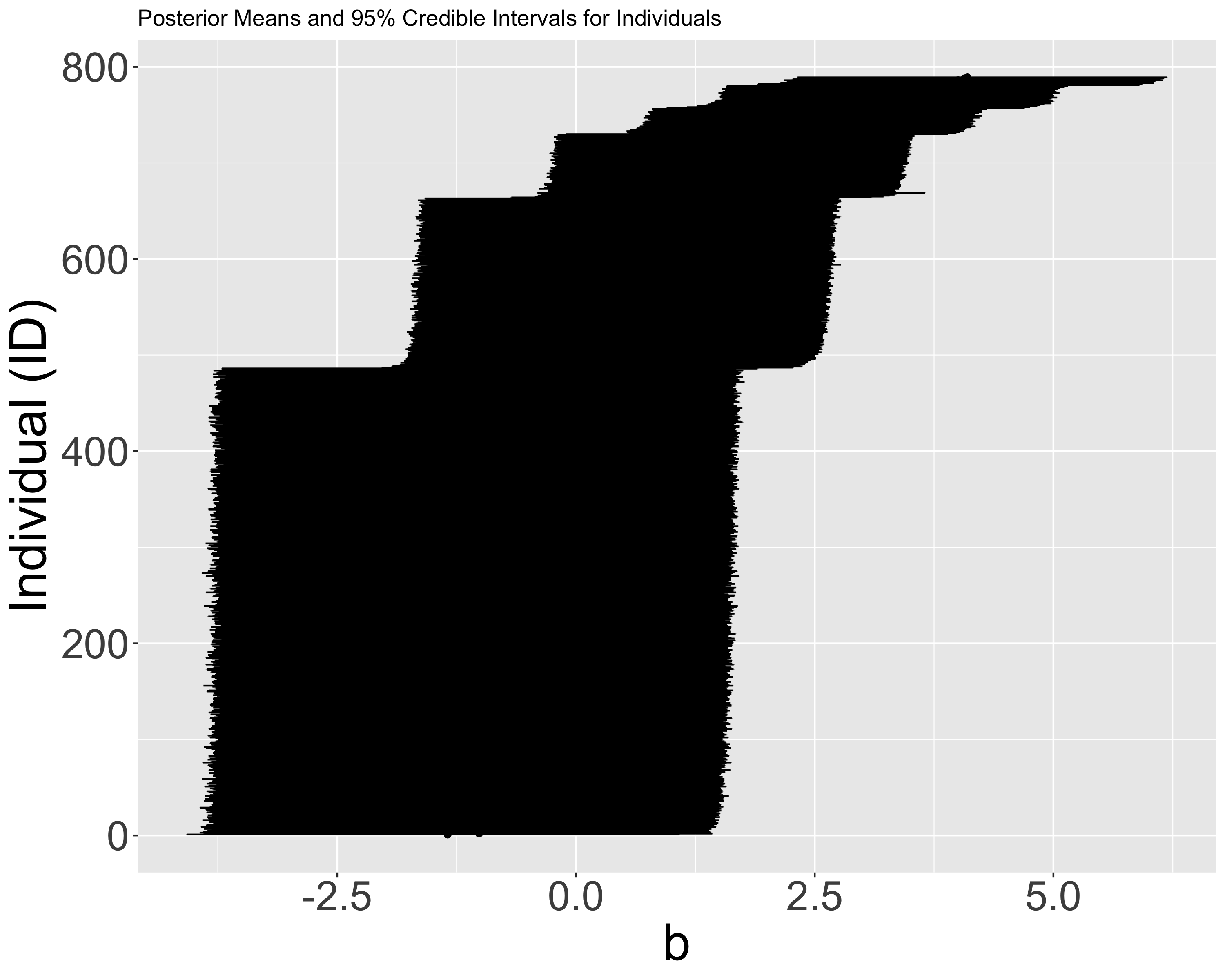
クラスタリングするだけでも精度が出そうだな.