備考は全て外し,LOX_Index と 判別式 も説明変数としては考慮しない.
| 64 |
2 |
54.2 |
86 |
2.6 |
22.8 |
2 |
438 |
13.0 |
5.8 |
24 |
23 |
70 |
243 |
70 |
96 |
93 |
135 |
-1 |
151 |
97 |
0 |
0 |
4 |
2 |
3 |
1 |
1 |
1 |
1 |
High |
| 48 |
2 |
52.7 |
75 |
2.2 |
22.1 |
2 |
452 |
14.0 |
5.5 |
23 |
18 |
25 |
282 |
64 |
91 |
97 |
171 |
-1 |
131 |
85 |
2 |
1 |
4 |
2 |
3 |
3 |
4 |
3 |
3 |
Normal |
| 53 |
2 |
64.8 |
55 |
2.8 |
26.0 |
2 |
464 |
13.9 |
5.5 |
16 |
14 |
19 |
262 |
106 |
99 |
69 |
171 |
-1 |
109 |
75 |
0 |
0 |
2 |
2 |
4 |
2 |
4 |
3 |
2 |
Normal |
| 46 |
1 |
76.3 |
136 |
3.8 |
26.0 |
2 |
553 |
16.3 |
5.9 |
17 |
16 |
56 |
221 |
148 |
107 |
50 |
139 |
-1 |
132 |
95 |
2 |
2 |
1 |
2 |
2 |
3 |
2 |
3 |
2 |
High |
| 65 |
1 |
72.4 |
58 |
2.3 |
26.2 |
2 |
520 |
16.4 |
5.4 |
16 |
10 |
28 |
201 |
97 |
90 |
77 |
104 |
-1 |
136 |
88 |
2 |
2 |
3 |
2 |
2 |
3 |
1 |
2 |
1 |
Normal |
| 30 |
1 |
59.5 |
46 |
2.2 |
19.5 |
2 |
555 |
16.0 |
5.2 |
18 |
18 |
26 |
185 |
169 |
89 |
49 |
103 |
-1 |
120 |
73 |
0 |
0 |
1 |
2 |
3 |
5 |
4 |
5 |
4 |
Normal |
[1] High Normal Normal High Normal Normal
Levels: High Normal
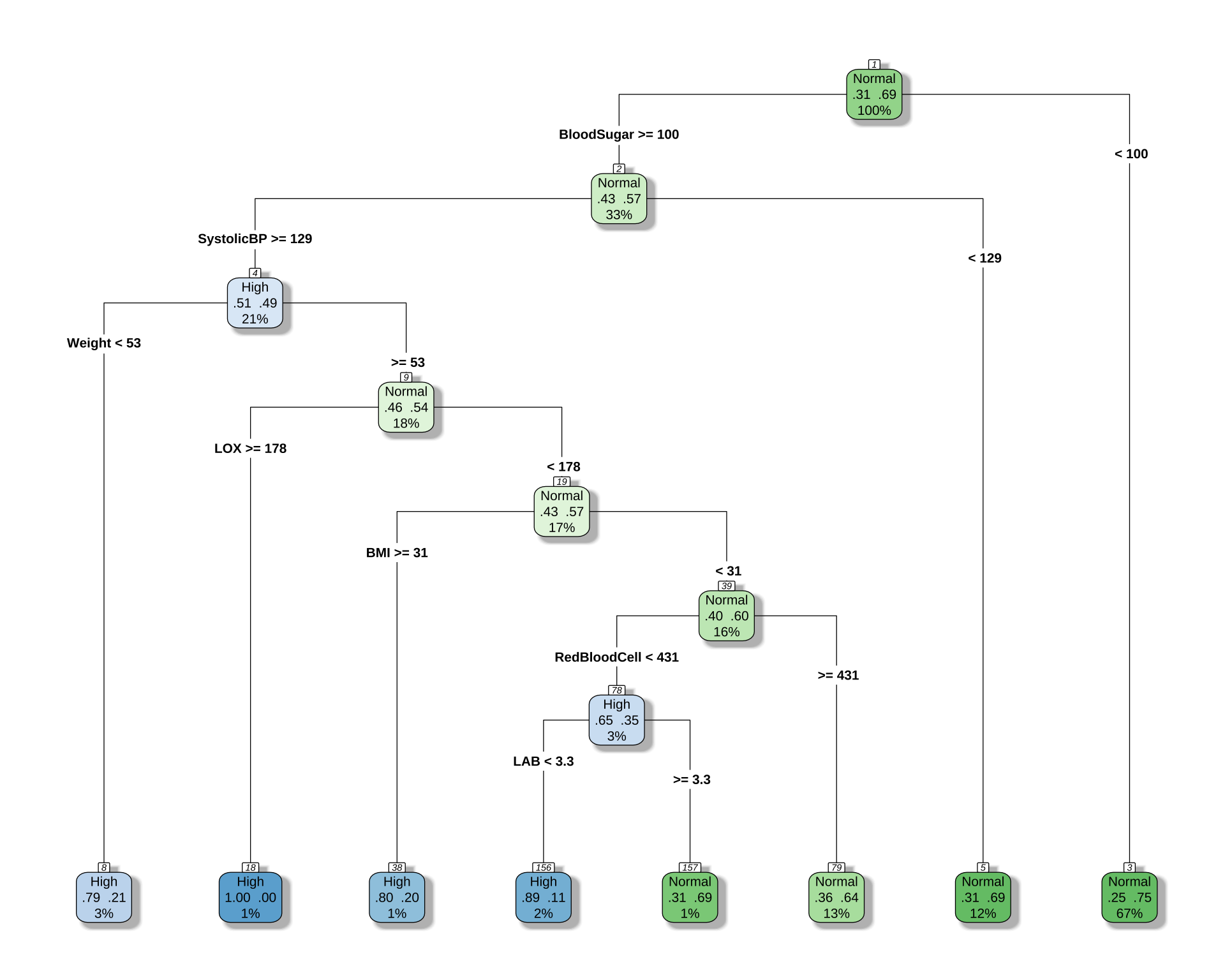
CART モデル
Warning: パッケージ 'rpart' はバージョン 4.3.3 の R の下で造られました
Warning: パッケージ 'rpart.plot' はバージョン 4.3.3 の R の下で造られました
# 目的変数が5クラスになっても、式は同じ
# rpartが自動で多クラス分類として扱ってくれる
cart_model <- rpart(
BP_level ~ ., # 目的変数を5クラスのものに変更
data = df_filtered,
method = "class",
control = rpart.control(cp = 0.01)
)
printcp(cart_model)
Classification tree:
rpart(formula = BP_level ~ ., data = df_filtered, method = "class",
control = rpart.control(cp = 0.01))
Variables actually used in tree construction:
[1] BloodSugar BMI LAB LOX RedBloodCell
[6] SystolicBP Weight
Root node error: 363/1162 = 0.31239
n= 1162
CP nsplit rel error xerror xstd
1 0.020202 0 1.00000 1.0000 0.043523
2 0.013774 6 0.86777 1.0220 0.043779
3 0.010000 7 0.85399 1.0303 0.043872
# 決定木を可視化
rpart.plot(
cart_model,
type = 4,
extra = 104, # 各ノードのクラス別サンプル数を表示
box.palette = "auto", # 色を自動で設定
shadow.col = "gray",
nn = TRUE
)
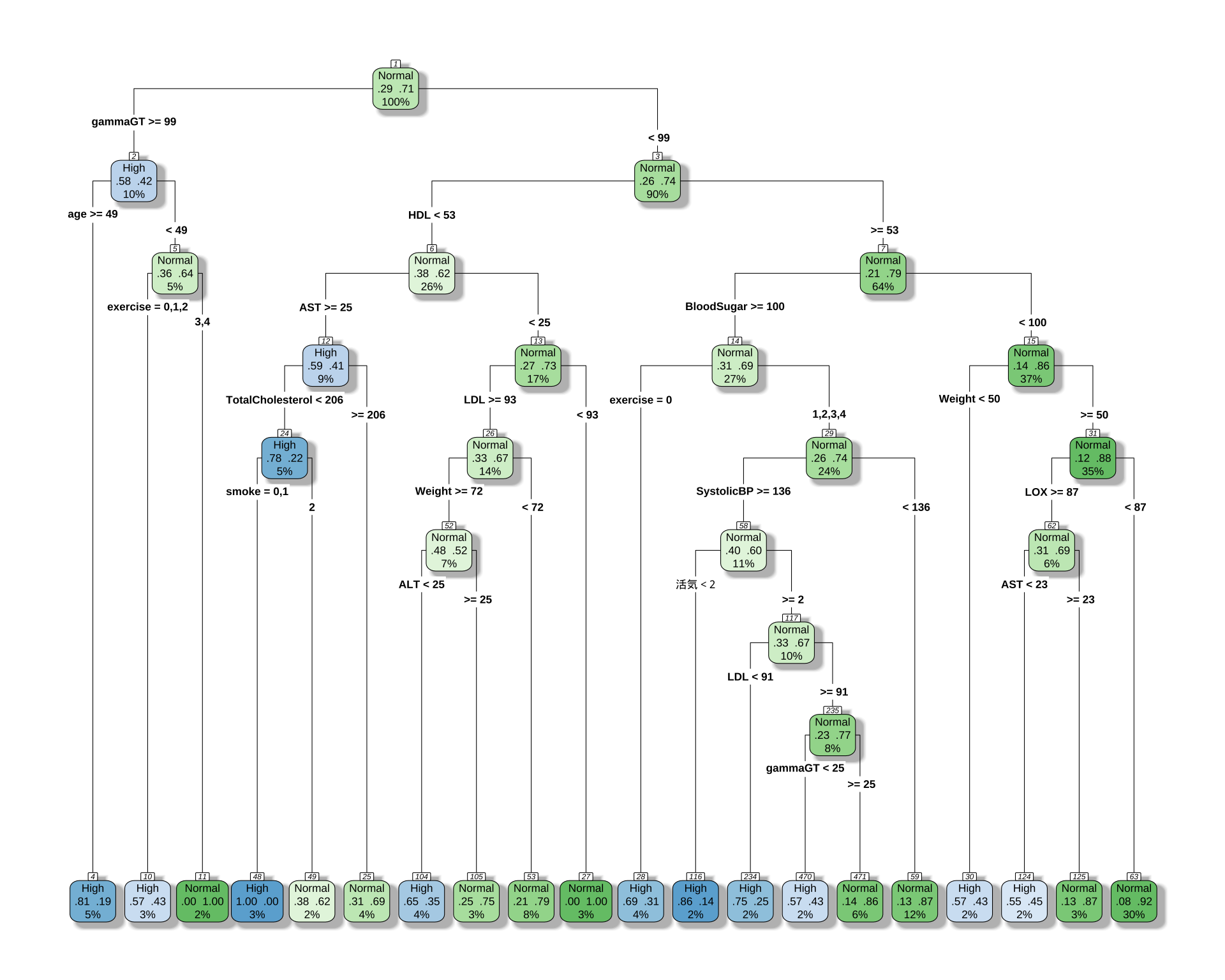
男女で違う CART モデルを推定する
男性の場合正答率が6割しか出ないが,女性の場合は8割出る.これは女性の方に C, E type がないためである.
男性の場合
「 BMI が小さくて多様性が3ならば A タイプ」というルールは変わらないようである.
library(rpart)
library(rpart.plot)
df_male <- df_filtered %>%
filter(sex == 1)
# 目的変数が5クラスになっても、式は同じ
# rpartが自動で多クラス分類として扱ってくれる
cart_model_5class <- rpart(
BP_level ~ ., # 目的変数を5クラスのものに変更
data = df_male,
method = "class",
control = rpart.control(cp = 0.0)
)
printcp(cart_model_5class)
Classification tree:
rpart(formula = BP_level ~ ., data = df_male, method = "class",
control = rpart.control(cp = 0))
Variables actually used in tree construction:
[1] age ALT AST BloodSugar
[5] exercise gammaGT HDL LDL
[9] LOX smoke SystolicBP TotalCholesterol
[13] Weight 活気
Root node error: 130/446 = 0.29148
n= 446
CP nsplit rel error xerror xstd
1 0.0538462 0 1.00000 1.0000 0.073825
2 0.0461538 1 0.94615 1.0231 0.074317
3 0.0269231 2 0.90000 1.0231 0.074317
4 0.0230769 5 0.80000 1.0154 0.074155
5 0.0192308 7 0.75385 1.0769 0.075390
6 0.0153846 10 0.68462 1.1077 0.075958
7 0.0128205 12 0.65385 1.1769 0.077120
8 0.0076923 15 0.61538 1.2231 0.077809
9 0.0038462 17 0.60000 1.2692 0.078430
10 0.0000000 19 0.59231 1.2462 0.078128
# 決定木を可視化
rpart.plot(
cart_model_5class,
type = 4,
extra = 104, # 各ノードのクラス別サンプル数を表示
box.palette = "auto", # 色を自動で設定
shadow.col = "gray",
nn = TRUE
)
# 予測値を取得
predictions <- predict(cart_model_5class, newdata = df_male, type = "class")
# 混同行列を作成
confusion_matrix <- table(Actual = df_male$BP_level, Predicted = predictions)
print(confusion_matrix)
Predicted
Actual High Normal
High 88 42
Normal 35 281
# 正答率を計算
accuracy <- sum(predictions == df_male$BP_level, na.rm = TRUE) / sum(!is.na(df_male$BP_level))
cat("正答率:", round(accuracy * 100, 2), "%\n")
58% の正解率が 71% になる.
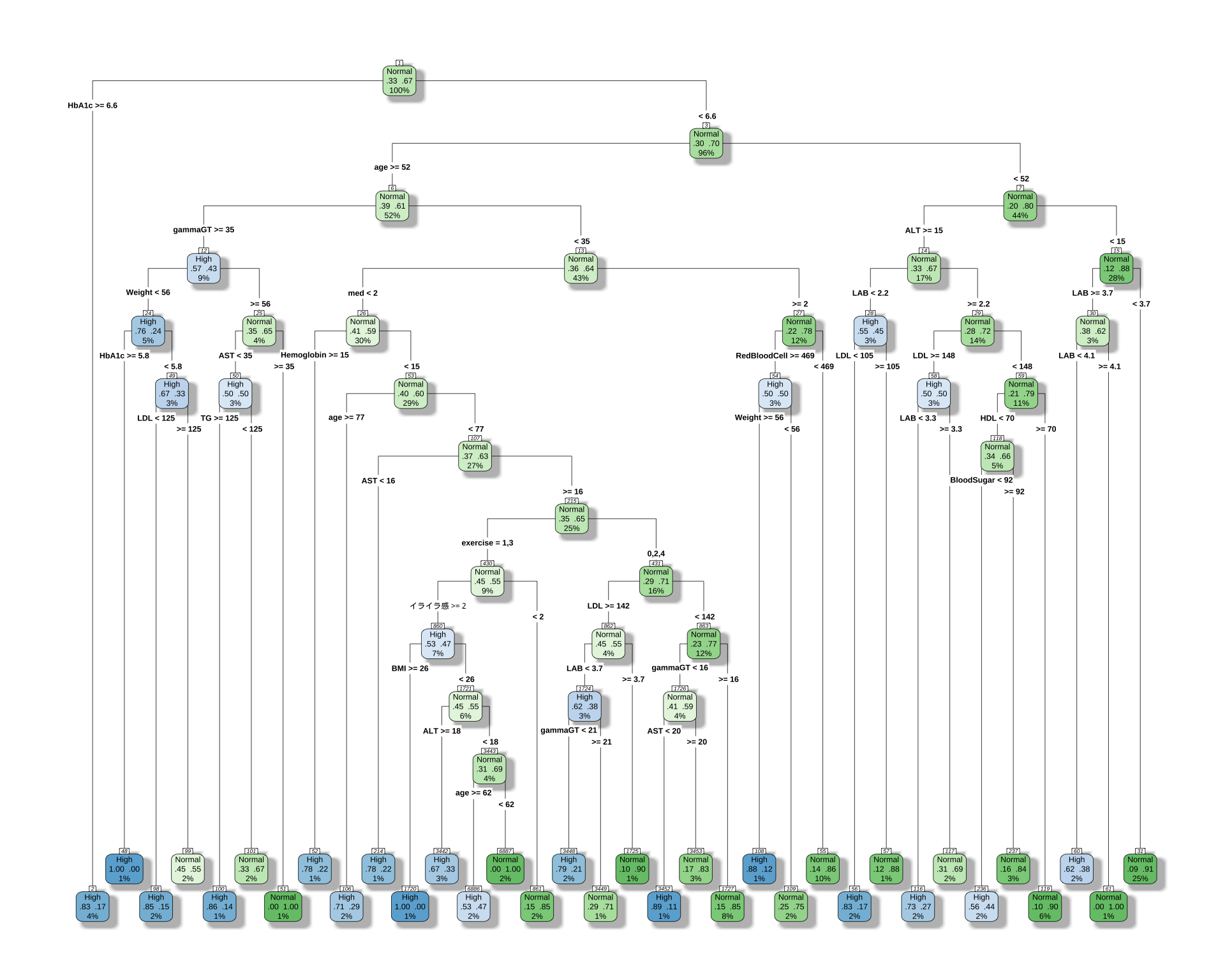
女性の場合
library(rpart)
library(rpart.plot)
df_female <- df_filtered %>%
filter(sex == 2)
# 目的変数が5クラスになっても、式は同じ
# rpartが自動で多クラス分類として扱ってくれる
cart_model_5class <- rpart(
BP_level ~ ., # 目的変数を5クラスのものに変更
data = df_female,
method = "class",
control = rpart.control(cp = 0.001)
)
printcp(cart_model_5class)
Classification tree:
rpart(formula = BP_level ~ ., data = df_female, method = "class",
control = rpart.control(cp = 0.001))
Variables actually used in tree construction:
[1] age ALT AST BloodSugar BMI
[6] exercise gammaGT HbA1c HDL Hemoglobin
[11] LAB LDL med RedBloodCell TG
[16] Weight イライラ感
Root node error: 233/716 = 0.32542
n= 716
CP nsplit rel error xerror xstd
1 0.0815451 0 1.00000 1.0000 0.053807
2 0.0257511 1 0.91845 1.0000 0.053807
3 0.0171674 4 0.84120 1.0601 0.054591
4 0.0139485 8 0.76824 1.0472 0.054432
5 0.0128755 12 0.71245 1.0558 0.054539
6 0.0114449 19 0.62232 1.0515 0.054486
7 0.0107296 22 0.58798 1.0858 0.054896
8 0.0064378 26 0.54506 1.0901 0.054945
9 0.0042918 28 0.53219 1.0987 0.055041
10 0.0021459 31 0.51931 1.0858 0.054896
11 0.0010000 33 0.51502 1.0901 0.054945
# 決定木を可視化
rpart.plot(
cart_model_5class,
type = 4,
extra = 104, # 各ノードのクラス別サンプル数を表示
box.palette = "auto", # 色を自動で設定
shadow.col = "gray",
nn = TRUE
)
# 予測値を取得
predictions <- predict(cart_model_5class, newdata = df_female, type = "class")
# 混同行列を作成
confusion_matrix <- table(Actual = df_female$BP_level, Predicted = predictions)
print(confusion_matrix)
Predicted
Actual High Normal
High 165 68
Normal 52 431
# 正答率を計算
accuracy <- sum(predictions == df_female$BP_level, na.rm = TRUE) / sum(!is.na(df_female$BP_level))
cat("正答率:", round(accuracy * 100, 2), "%\n")
少ない変数でやったときはタイプ B 259 人から,261 人になっただけだったが,今回はもう少し増えるようだ.